Mise à jour importante:
Le projet de Résistance aux antimicrobiens 2 de l'IRDG a été renommé Résistance aux antimicrobiens - Une seule santé. Ce changement reflète notre engagement à adopter une approche globale de la résistance aux antimicrobiens, qui intègre la santé humaine, animale et environnementale.
Période de financement : 2022–2027
Responsables : Tim McAllister, Agriculture et Agroalimentaire Canada, et Catherine Carrillo, Agence d'inspection des aliments
Financement total de l'IRDG : 9 800 000 $
La résistance aux médicaments antimicrobiens, aussi appelée résistance aux antimicrobiens (RAM), constitue un défi important en matière de santé publique. En particulier, le développement de la résistance aux antimicrobiens par des agents pathogènes bactériens et fongiques qui étaient auparavant sensibles est une menace importante pour la santé humaine et animale. Reconnaissant la nature complexe du problème de la RAM, l'Initiative de recherche et développement en génomique (IRDG) finance un deuxième projet interministériel sur la résistance aux antimicrobiens, RAM-SS.
Le premier projet de RAM de l'IRDG était axé sur l'examen de la RAM dans le contexte des systèmes de production agricole. Grâce aux travaux réalisés dans le cadre de ce projet, les chercheurs ont acquis une meilleure connaissance des processus essentiels qui contribuent à l'apparition de la RAM dans les systèmes de production d'aliments et ont déterminé les principales voies d'exposition par lesquelles les bactéries RAM d'origine agricole se transmettent aux humains.
En s'appuyant sur ces résultats, le projet RAM-SS permettra de poursuivre l'étude des risques associés à l'apparition et à la transmission de la RAM. Les milieux fédéraux de la génomique et de la réglementation qui s'intéressent à la RAM ont établi les objectifs généraux du projet et ils ont défini que le modèle de gestion des questions liées aux antimicrobiens touchant la santé humaine, animale et environnementale (approche « Une santé ») constituait la meilleure approche pour la création d'une synergie entre les ministères collaborateurs. L'élargissement du champ de ce projet permettra de s'attaquer à la nature interconnectée des voies de transmission de la RAM, qui touchent des domaines de responsabilité propres au mandat. Bien que le projet de l'IRDG RAM-SS permette de poursuivre l'étude de la transmission de la RAM dans le contexte des systèmes de production agroalimentaire, le champ de ce projet portera également sur les systèmes naturels et de traitement des eaux usées, les pêches et les milieux de soins de santé.
Contexte
Au total, 33 scientifiques et leurs équipes provenant de 6 ministères et organismes scientifiques fédéraux contribuent au projet RAM-SS. Ils tirent parti des programmes fédéraux d'échantillonnage et de dépôts d'échantillons existants pour cartographier le développement et la transmission de la résistance aux antimicrobiens par les soins de santé, les animaux, les végétaux, les eaux usées et l'environnement à l'aide d'approches génomiques, dont la métagénomique. Le séquençage du génome entier et l'épidémiologie génomique. L'équipe élabore également des outils et des méthodes d'analyse de calibre mondial pour l'analyse génomique. Non seulement ces outils et méthodes sont partagés dans le cadre du projet, ils sont aussi mis à la disposition des scientifiques universitaires et de l'industrie, conformément à un engagement envers la science ouverte. De même, l'équipe publie et communique régulièrement les résultats de ses recherches, notamment en les mettant à disposition dans des bases de données universitaires pour assurer une diffusion rapide et large des connaissances obtenues grâce au projet.
Le projet entend élaborer et mettre en application des solutions pratiques pour contrôler et réduire la RAM en étudiant les relations entre la RAM et le recours aux antimicrobiens et en analysant la migration de la RAM dans différents nœuds du continuum « Une santé » afin de mieux comprendre les modes de transmission. Les connaissances obtenues serviront à déterminer les points chauds et les points d'intervention et à étayer les stratégies d'atténuation fondées sur des données probantes, dont les soins de santé éclairés et les pratiques agroalimentaires, les règlements et les politiques ainsi que l'éducation des consommateurs pour protéger les populations vulnérables. L'objectif primordial est de préserver l'efficacité des antimicrobiens sur lesquels la population canadienne compte chaque jour.
Figure 1 : Le projet « Une seule santé » sur la RAM

Figure 1 : Le projet « Une seule santé » sur la RAM - Version textuelle
Le projet « Une seule santé » sur la résistance aux antimicrobiens (RAM) est organisé en six thèmes. Chaque thème mobilise un ensemble de ministères et d'agences fédérales à vocation scientifique qui collaborent pour mener à bien des travaux dans différents domaines :
- Évaluer la RAM et sa diffusion dans et entre différents environnements, tels que la production alimentaire terrestre et aquatique, l'agriculture et les environnements naturels terrestres et aquatiques. Collaborateurs : Agriculture et Agroalimentaire Canada (responsable), l'Agence canadienne d'inspection des aliments, Pêches et Océans Canada, Environnement et Changement climatique Canada et l'Agence de santé publique du Canada.
- Étudier la RAM dans les établissements de soins de santé, les communautés et les populations vulnérables. Collaborateur : Agence de la santé publique du Canada.
- Atténuer le développement et la transmission de la RAM tout au long du continuum « Une seule santé » : de la ferme à la table et à la clinique. Collaborateurs : Agriculture et Agroalimentaire Canada (responsable) et Pêches et Océans Canada.
- Comparer les pathogènes résistants aux antimicrobiens à l'échelle du continuum « Une seule santé ». Collaborateurs : Agriculture et Agroalimentaire Canada (responsable), Agence canadienne d'inspection des aliments, Santé Canada et Agence de santé publique du Canada.
- Tirer parti de la génomique (étude des gènes et de l'ADN) pour évaluer les risques. Collaborateurs : Agriculture et Agroalimentaire Canada, Agence canadienne d'inspection des aliments, Pêches et Océans Canada, Environnement et Changement climatique Canada, Santé Canada, CNRC et Agence de santé publique du Canada.
- Détecter la RAM en temps réel à l'échelle du continuum « Une seule santé ». Tous les collaborateurs : Agriculture et Agroalimentaire Canada, Agence canadienne d'inspection des aliments, Pêches et Océans Canada, Environnement et Changement climatique Canada, Santé Canada, CNRC et Agence de santé publique du Canada.
Faits saillants
- Collaboration quinquennale
- Trente-trois (33) scientifiques et leurs équipes
- Six (6) ministères et organismes fédéraux
Figure 2 : Les interactions et les voies de transmission de la RAM
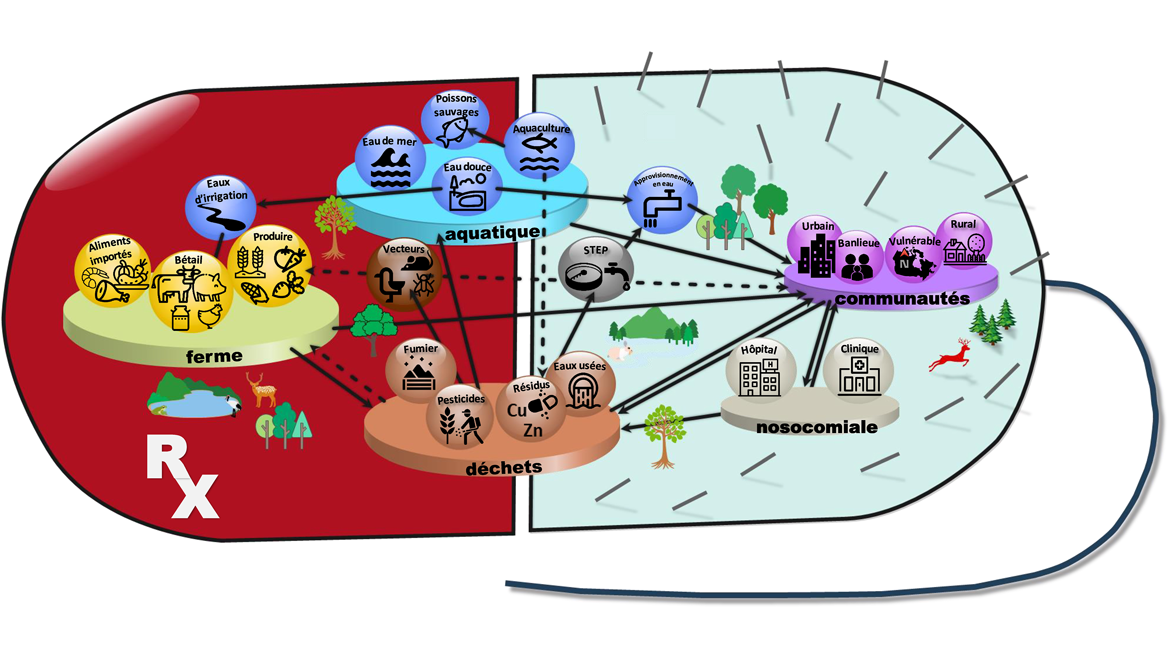
Figure 2 : Les interactions et les voies de transmission de la RAM - Version textuelle
Représentation d'une cellule bactérienne prenant la forme d'une capsule d'antibiotique, montrant les interactions entre les voies possibles de transmission de la résistance aux antimicrobiens entre des environnements aquatiques et terrestres. Sur cet arrière-plan, cinq zones environnementales sont superposées ainsi que les voies de transmission des bactéries résistantes aux antimicrobiens entre elles, toutes entourées de représentations de la nature telles que des arbres, des cerfs, des oiseaux, des lacs et des montagnes. De nombreuses flèches bidirectionnelles pleines et pointillées relient tous les environnements dans un schéma élaboré et entrecroisé, indiquant des interactions complexes entre eux. Chaque environnement présente des exemples de ses composants sous forme d'icônes accompagnées de texte :
5 environnements
- Aquatique : Aquaculture, eaux douces, eaux océaniques et poissons sauvages
- Communautés : Urbaines, périurbaines, vulnérables et rurales
- Nosocomial : Hôpital et clinique
- Déchets : Fumier, pesticides, eaux usées et résidus tels que le zinc et le cuivre.
- Exploitation agricole : Bétail, fruits et légumes, eaux d'irrigation et aliments importés
Mise en évidence des voies de transmission entre les environnements
- Approvisionnement en eau
- Eau d'irrigation
- Eaux usées
- Vecteurs animaux : mammifères, insectes et oiseaux
Objectif
Déterminer les cibles d'intervention à l'aide d'une approche « Une santé » en cartographiant le développement et la transmission de la résistance aux antimicrobiens dans les milieux de soins de santé, et par les animaux, les végétaux, les eaux usées et l'environnement.
Retombées positives attendues
- Détermination des points d'intervention, menant à l'amélioration des pratiques de surveillance et des stratégies d'intervention
- Réduction de la transmission de la RAM pour préserver l'efficacité des antimicrobiens sur lesquels la population canadienne compte chaque jour
Domaines d'intérêt
Évaluation des facteurs, des réservoirs et de la transmission de la RAM dans les matrices environnementales
Ce domaine vise à tirer parti de l'infrastructure scientifique existante, dont le séquençage de prochaine génération et les outils bio-informatiques, qui évoluent rapidement, ainsi que de l'expertise de plusieurs organismes pour étudier l'abondance et la distribution des gènes de la RAM au sein de différentes communautés microbiennes (résistomes) tout au long du continuum « Une santé ».
Soins de santé et collectivités canadiennes, dont les populations vulnérables
Ce domaine vise à déterminer les points chauds et les points d'intervention en matière de RAM afin de réduire la transmission de la RAM entre les établissements de soins de santé, et les autres populations à risque et les communautés.
Atténuation du développement et de la transmission de la RAM dans le continuum de la ferme à la table et à la clinique
Ce domaine vise à trouver des solutions pour réduire le recours aux antimicrobiens dans les industries de production d'aliments en optimisant les pratiques existantes et en introduisant de nouvelles méthodes qui réduisent le besoin d'antimicrobiens. Cette recherche appliquée permettra de renforcer les approches de gestion qui peuvent avoir une incidence sur la communauté bactérienne dans la production d'animaux destinés à l'alimentation.
Comparaison phylogénomique des agents pathogènes présentant une RAM et provenant de milieux agricoles, environnementaux, alimentaires et cliniques du pays
Ce domaine vise à tirer parti de l'expertise et des ressources de plusieurs ministères pour étudier la transmission de la RAM entre les communautés le long du continuum de la ferme à la table et à la clinique. Les résultats de la recherche permettront de cartographier les voies de transmission et de déterminer les points chauds potentiels et les espèces bactériennes à risque élevé, ce qui guidera l'élaboration de stratégies d'intervention éclairées.
Mise à profit de la génomique dans la science des risques
Ce domaine vise à intégrer les données de recherche du projet afin d'examiner les facteurs qui contribuent à l'augmentation des risques associés à la RAM pour la santé humaine, animale et environnementale au moyen de deux avenues :
- Combler certaines lacunes dans les connaissances sur les processus qui pourraient contribuer à la dissémination des bactéries résistantes aux antibiotiques, de gènes de résistance aux antibiotiques et d'éléments génétiques mobiles.
- Élaborer des produits de la science des risques (p. ex., évaluations des risques, modèles de risque) éclairés par les données génomiques obtenues grâce au projet.
Détection rapide et communication des résultats de la RAM dans l'ensemble du continuum « Une santé »
Ce domaine vise à permettre à tous les participants d'accéder facilement aux souches, aux réactifs et aux données et aux renseignements et interpréter ces deux derniers avec précision.
Principaux produits livrables
- Établissement de points chauds de la RAM dans l'environnement et des principaux facteurs qui seront à prioriser aux fins des efforts d'atténuation
- Grâce à l'épidémiologie moléculaire, établissement de voies de transfert (« reliant les points ») entre la RAM dans les isolats cliniques prélevés chez l'humain et les bactéries présentant une RAM provenant de systèmes de production agricole, des communautés humaines et de l'environnement
- Identification des vecteurs de la dissémination de la RAM dans les milieux naturels, et les milieux terrestres et aquatiques touchés par l'activité humaine.
- Établissement des principales sources de RAM dans les populations humaines et des voies de transmission entre les collectivités, les établissements de soins de santé et d'autres populations à risque (p. ex. établissements de soins de longue durée, communautés nordiques et autochtones, établissements correctionnels)
- Inclusion de données génomiques dans les analyses des risques et modélisation des risques afin de déterminer d'autres approches nouvelles pour atténuer la RAM dans l'ensemble du continuum « Une santé »
- Recensement d'études prospectives pour évaluer les effets des fongicides et des antibiotiques utilisés dans la production végétale sur le développement de la RAM chez les bactéries et les champignons de l'environnement, ce qui fournira aux autorités réglementaires des données pertinentes pour les évaluations des risques pour l'environnement.
- Efficacité des stratégies d'intervention visant à réduire le recours aux antimicrobiens dans les systèmes de production d'aliments, à valider dans des conditions commerciales
- Évaluation de la probabilité d'exposition à la RAM selon les pratiques de production d'aliments utilisées par les producteurs nationaux et internationaux, en fonction de la surveillance des aliments canadiens et importés (viande crue et prête à manger, de même que des produits frais et congelés)
- Données génomiques pour les outils d'évaluation épidémiologique et des risques visant à fournir des données probantes plus solides pour éclairer la prise de décisions dans divers contextes, dont les systèmes de production d'aliments terrestres et aquatiques, la gestion de l'environnement et les milieux de soins de santé.
Participants financés
Collaborateurs
- BC Centre for Aquatic Health Sciences (en anglais seulement)
- Réseau laitier canadien
- Université Carleton (en anglais seulement)
- Université Dalhousie (en anglais seulement)
- Health Sciences Centre Winnipeg (en anglais seulement)
- Université McGill
- Université McMaster (en anglais seulement)
- Hôpital Mount Sinai (en anglais seulement)
- Université Simon Fraser (en anglais seulement)
- Université de l'Alberta (en anglais seulement)
- Université de la Colombie-Britannique (en anglais seulement)
- Université de Cambridge (en anglais seulement)
- Université de Guelph (en anglais seulement)
- Université de Lethbridge (en anglais seulement)
- Université de l'Île-du-Prince-Édouard (en anglais seulement)
- Université de la Saskatchewan (en anglais seulement)
- Université du Texas (en anglais seulement)
- Université de Waterloo (en anglais seulement)
- Université de Windsor (en anglais seulement)
Publications
- Bradford, L., L. Yao, C. Anastasiadis, A. Cooper, B. Blais, A. Deckert, R. Reid-Smith, C. Lau, M. S. Diarra, C. Carrillo, and A. Wong, 2024. Limit of detection of Salmonella ser. Enteritidis using culture-based versus culture-independent diagnostic approaches. Microbiology Spectrum, https://doi.10.1128/spectrum.01027-24 (en anglais seulement).
- Brown LP, Murray R, Scott A, Tien YC, Lau CHF, Tai V, Topp E. 2022. Responses of the soil bacterial community, resistome, and mobilome to a decade of annual exposure to macrolide antibiotics. Appl Environ Microbiol 6;88(8):e0031622. https://doi.org/10.1128/aem.00316-22 (en anglais seulement)
- Chongwu Y, Diarra MS, Rehman MA, Li L, Yu H, Yin X, Aslam M, CCarrillo CD, Yang C, Gong J. 2023. Virulence potential of antimicrobial resistant extraintestinal pathogenic E. coli from retail poultry meat in a Caenorhabditis elegans model. J. Food Protect. 86 (2023) 100008. https://doi.org/10.1016/j.jfp.2022.11.001 (en anglais seulement)
- Cooper AL, Low A, Wong A, Tamber S, Blais BW, Carrillo CD. 2024. Modeling the limits of detection for antimicrobial resistance genes in agri-food samples: a comparative analysis of bioinformatics tools. BMC Microbiol 24:31. https://doi.org/10.1186/s12866-023-03148-6 (en anglais seulement)
- Cooper AL, Wong A, Tamber S, Blais BW, Carrillo CD. 2024. Analysis of Antimicrobial Resistance in Bacterial Pathogens Recovered from Food and Human Sources: Insights from 639,087 Bacterial Whole-Genome Sequences in the NCBI Pathogen Detection Database. Microorganisms. 12(4):709. https://doi.org/10.3390/microorganisms12040709 (en anglais seulement)
- Das Q, Hasted TL, Lepp D, Yin X, Tang J, Chalmers G, Ross K, Boerlin P, Diarra MS. 2023. Transcriptional profiling of extraintestinal Escherichia coli exposed to cranberry pomace extracts alone or in combination with ceftriaxone. Front. Sustain. Food Syst. 6:957099. http://doi.org/10.3389/fsufs.2022.957099 (en anglais seulement)
- Demars M, McDowell T, Renaud J, Scott A, Fruci M, Topp E. 2024. Persistence and evidence for accelerated biodegradation of streptomycin in agricultural soils, Science of The Total Environment, 2024,172502,ISSN 0048-9697, https://doi.org/10.1016/j.scitotenv.2024.172502 (en anglais seulement)
- Diarra MS, Zhao X, Butaye PR. 2023. Antimicrobial use, antimicrobial resistance, and the microbiome in animals: volume II. Front. Vet. Sci. Volume 10. https://doi.org/10.3389/fvets.2023.1149010 (en anglais seulement)
- Diarra MS, Zhao X, Butaye PR, eds. 2023. Antimicrobial use, antimicrobial resistance, and the microbiome in animals, volume II. Lausanne: Frontiers Media SA. https://doi.org/10.3389/978-2-83251-839-7 (en anglais seulement)
- Flint A, Cooper A, Rao M, Weedmark K, Carrillo C, Tamber S. 2023. Targeted metagenomics using bait-capture to detect antibiotic resistance genes in retail meat and seafood. Front Microbiol. 2023 Jul 13;14:1188872. https://doi.org/10.3389/fmicb.2023.1188872 (en anglais seulement)
- Flint A, Laidlaw A, Li L, Raitt C, Rao M, Cooper A, Weedmark K, Carrillo C, Tamber S. 2022. Choice of DNA extraction method affects detection of bacterial taxa from retail chicken breast. BMC Microbiology. 22:230. https://doi.org/10.1186/s12866-022-02650-7 (en anglais seulement)
- Flores-Vargas G, Korber DR, Bergsveinson J. 2023. Sub-MIC antibiotics influence the microbiome, resistome and structure of riverine biofilm communities. Front. Microbiol. Sec. Aquatic Microbiology 31. https://doi.org/10.3389/fmicb.2023.1194952 (en anglais seulement)
- George PBL, Rossi F, St-Germain MW, Amato P, Badard T, Bergeron MG, Boissinot M, Charette SJ, Coleman BL, Corbeil J, Culley AI, Gaucher ML, Girard M, Godbout S, Kirychuk SP, Marette A, McGeer A, O'Shaughnessy PT, Parmley EJ, Simard S, Reid-Smith RJ, Topp E, Trudel L, Yao M, Brassard P, Delort AM, Larios AD, Létourneau V, Paquet VE, Pedneau MH, Pic É, Thompson B, Veillette M, Thaler M, Scapino I, Lebeuf M, Baghdadi M, Castillo Toro A, Cayouette AB, Dubois MJ, Durocher AF, Girard SB, Diaz AKC, Khalloufi A, Leclerc S, Lemieux J, Maldonado MP, Pilon G, Murphy CP, Notling CA, Ofori-Darko D, Provencher J, Richer-Fortin A, Turgeon N, Duchaine C. 2022. Antimicrobial Resistance in the Environment: Towards Elucidating the Roles of Bioaerosols in Transmission and Detection of Antibacterial Resistance Genes. Antibiotics (Basel). 11(7):974. https://doi.org/10.3390/antibiotics11070974 (en anglais seulement)
- Hetman BM, Pearl DL, Barker DOR, Robertson J, Nash JHE, Reid-Smith R, Agunos A, Carrillo C, Topp E, Van Domselaar G, Parmley EJ, Bharat A, Mulvey M, Allen V, Taboada EN. 2022. Combining analytical epidemiology and genomic surveillance to identify risk factors associated with the spread of antimicrobial resistance in Salmonella enterica subsp. enterica serovar Heidelberg. Microb Genom. 8(11):mgen000891. https://doi.org/10.1099/mgen.0.000891 (en anglais seulement)
- Isada MJ, Reist M, MacKinnon MC, Uhland FC, Young KM, Gibbens K, Parmley EJ, Carson CA. 2022. Characterisation of burden of illness measures associated with human (Fluoro)quinolone-resistant Campylobacter spp. infections - a scoping review. Epidemiol Infect. 150:e205. https://doi.org/10.1017/S095026882200139X (en anglais seulement)
- Johnson L, Dufour S, Smith DDN, Manning A, Bulbul A, Binette S, Hamoutene D. 2023. Descriptive analyses of microbial communities in marine sediment microcosms spiked with fish wastes, emamectin benzoate, and oxytetracycline. Ecotoxicology and Environmental Safety, Volume 268, 115683, https://doi.org/10.1016/j.ecoenv.2023.115683 (en anglais seulement)
- Jonah L, Hamoutene D, Kingsbury M, Johnson L, Fenton AJ. 2024. A data compilation of antibiotic treatments in Canadian finfish aquaculture from 2016 to 2021 and the cumulative usage of antibiotics and antiparasitic drugs at marine sites. Environmental Reviews. https://doi.org/10.1139/er-2023-0124 (en anglais seulement)
- Kang H, Wang Q, Yu H, Guo Q, Weber L, Wu W, Lepp D, Cui SW, Diarra MS, Liu H, Shao S, Gong J. 2024. Validating the use of a newly developed cinnamaldehyde product in commercial broiler production. Poultry Science. https://doi.org/10.1016/j.psj.2024.103625 (en anglais seulement)
- Kang M, Charron P, Hoover E, Guan J, Firth I, Naushad I, and Huang H (correspondence). 2023. Complete genome sequence of a Canadian strain of Escherichia coli with multiple metal and antimicrobial resistance genes isolated from municipal biosolids. Microbiology Resources Announcement, ASM. 2023. Volume 12, Issue 5, e00083-23. https://doi.org/10.1128/mra.00083-23 (en anglais seulement)
- Kang M, Charron P, Hoover E, Huang H. 2024. Complete genome sequences of a Canadian strain of enteroaggregative Escherichia coli (EAEC) with multiple metals and antimicrobial resistance genes isolated from municipal waste-activated sludge. Microbiol Resour Announc 13:e01242-23. https://doi.org/10.1128/mra.01242-23 (en anglais seulement)
- Kang M, Naushad S, Hartke A, Firth I, Madey E, Ogunremi D, Huang H. 2022. Antibiotic resistomes and microbial communities in biosolid fertilizers collected from two Canadian wastewater treatment plants in a 10-years interval-potential risks to food chains? Front. Food. Sci. Technol. 2:894671. https://doi.org/10.3389/frfst.2022.894671 (en anglais seulement)
- Kujat Choy S, Neumann EM, Romero-Barrios P, Tamber S. 2024. Contribution of Food to the Human Health Burden of Antimicrobial Resistance. Foodborne Pathog Dis. 21(2):71-82. https://doi.org/10.1089/fpd.2023.0099 (en anglais seulement)
- Lau CHF, Capitani S, Tien YC. et al. 2024. Dynamic effects of black soldier fly larvae meal on the cecal bacterial microbiota and prevalence of selected antimicrobial resistant determinants in broiler chickens. Anim. microbiome 6:6. https://doi.org/10.1186/s42523-024-00293-9 (en anglais seulement)
- Lee C, Polo RO, Zaheer R, Van Domselaar G, Zovoilis A, McAllister T A. 2023. Evaluation of metagenomic assembly methods for the detection and characterization of antimicrobial resistance determinants and associated mobilizable elements. Journal of microbiological methods, 213, 106815. https://doi.org/10.1016/j.mimet.2023.106815 (en anglais seulement)
- Lin, H. M. S. Diarra, G. Jia, X. Zhao. 2024. Detection in Salmonella from poultry: investigating the potential horizontal transfer of antimicrobial resistance and virulence genes. Poultry Science # PSJ-D-24-02029R1.
- Lee C, Zaheer R, Munns K, Holman DB, Van Domselaar G, Zovoilis A, McAllister TA. 2023. Effect of Antimicrobial Use in Conventional Versus Natural Cattle Feedlots on the Microbiome and Resistome. Microorganisms, 11(12), 2982. https://doi.org/10.3390/microorganisms11122982 (en anglais seulement)
- Liu X, Floate KD,Gorzelak MA, Holman DB, Hrycauk S, Kubota H, Lupwayi N, Neilson JAD, Ortega PR, Petri RM, Tran L, Wang H, Wilches D, Yang X, Zorz J, Guarna MM. 2023. Prairie Agroecosystems: Interconnected Microbiomes of Livestock, Soil and Insects. Agriculture. 13:326. https://doi.org/10.3390/agriculture13020326 (en anglais seulement)
- Loest D, Uhland FC, Young KM, Li XZ, Mulvey MR, Reid-Smith R, Sherk LM, Carson CA. 2022. Carbapenem-resistant Escherichia coli from shrimp and salmon available for purchase by consumers in Canada: a risk profile using the Codex framework. Epidemiol Infect. 150:e148. https://doi.org/10.1017/S0950268822001030 (en anglais seulement)
- Lorenc, N., Leadbeater, S., Wang, J., Ronholm, J., Liu, X. 2024. A pilot study on the effects of in-feed probiotic Lactobacillus rhamnosus ATCC 53103 (LGG) on vaccinated Atlantic salmon (Salmo salar): Microbiomes and Aeromonas salmonicida challenge resilience. Can J Microbiol.
- Mak PHW, Rehman MA, Kiarie EG, Topp E, Diarra MS. 2022. Production systems and important antimicrobial resistant‑pathogenic bacteria in poultry: a review. Journal of Animal Science and Biotechnology. 13:148 https://doi.org/10.1186/s40104-022-00786-0 (en anglais seulement)
- McMahon TC, Kingombe CB, Mathews A, Seyer K, Wong A, Blais BW, Carrillo CD. 2022. Microbial Antagonism in Food-Enrichment Culture: Inhibition of Shiga Toxin-Producing Escherichia coli and Shigella Species. Frontiers in Microbiology 13: 880043. https://doi.org/10.3389/fmicb.2022.880043 (en anglais seulement)
- Muhammad B, Diarra MS, Islam MdR, Lepp D, Mastin-Wood RE, Topp E, Bittman S, Zhao X. 2022. Effects of litter from antimicrobial-fed broiler chickens on soil bacterial community structure and diversity. Can. J. Microbiol. 00: 1–11. https://doi.org/10.1139/cjm-2022-0086 (en anglais seulement)
- Nagarajan A, Goyette B, Raghavan V, Poulin-Laprade D, Rajagopal R. 2024. Integrating Anaerobic Digestion with Struvite Production for Enhanced Nutrient Recovery, Pathogen Reduction, and Circularity in Manure Management, Journal of Sustainable Agriculture and Environment, https://doi.org/10.1002/sae2.70018 (en anglais seulement)
- Oladeinde, A., K. Cook, A. Rehman, C. D. Carrillo, R. Woyda, C. Wiersma, Z. Abdo, J. Johnson Anna M. Bosch, M. Rothrock Jnr, and M. S. Diarra. 2024. Survival of Antimicrobial Resistant Salmonella Heidelberg Inoculated into Microcosms of Fresh Pine Wood Shavings for Broiler Litter. Can. J Microbiol. https://doi.10.1139/cjm-2024-0088 (en anglais seulement).
- Phillips C, Chapman B, Agunos A, Carson CA, Parmley EJ, Reid-Smith RJ, Smith BA, Murphy CP. 2022. A scoping review of factors potentially linked with antimicrobial-resistant bacteria from turkeys (iAM.AMR Project). Epidemiol Infect. 150:e153. https://doi.org/10.1017/S0950268822001224 (en anglais seulement)
- Primeau CA, Bharat A, Janecko N, Carson CA, Mulvey M, Reid-Smith R, McEwen S, McWhirter JE, Parmley EJ. 2022. Integrated surveillance of extended-spectrum beta-lactamase (ESBL)-producing Salmonella and Escherichia coli from humans and animal species raised for human consumption in Canada from 2012 to 2017. Epidemiol Infect. 151:e14. https://doi.org/10.1017/S0950268822001509 (en anglais seulement)
- Robertson J, Schonfeld J, Bessonov K, Bastedo P, Nash JHE. 2023. A global survey of Salmonella plasmids and their associations with antimicrobial resistance. Microbial Genomics 9, 001002. https://doi.org/10.1099/mgen.0.001002 (en anglais seulement)
- Scott A, Murray R, Tien YC, Topp E. 2022. Contamination of hay and haylage with enteric bacteria and selected antibiotic resistance genes following fertilization with dairy manure or biosolids. Can J Microbiol. 68(4):249-257. https://doi.org/10.1139/cjm-2021-0326 (en anglais seulement)
- Shay JA, Haniford LSE, Cooper A, Carrillo CD, Blais BW, Lau, CH. 2023. Exploiting a targeted resistome sequencing approach in assessing antimicrobial resistance in retail foods. Environ. Microbiome 18(1):25. https://doi.org/10.1186/s40793-023-00482-0 (en anglais seulement)
- • St-Laurent, R.E., Vincent, A. T., Paquet, V. E., Leduc, G. R., Lorenc, N.; Ronholm, J.; Liu, X.; Charette, S.J. 2024. Characterization of Aeromonas salmonicida mesophilic isolates from Alberta (Canada) allowing the development of a more sensitive Dictyostelium discoideum predation test. FEMS Microbiology. https://doi.org/10.1093/femsle/fnae078 (en anglais seulement).
- Strong KM, Marasco KL, Invik J, Ganshorn H, Reid-Smith RJ, Waldner CL, Otto SJG, Kastelic JP, Checkley SL. 2023. Factors associated with antimicrobial resistant enterococci in Canadian beef cattle: a scoping review. 2023. Front Vet Sci. 10:1155772. https://doi.org/10.3389/fvets.2023.1155772 (en anglais seulement)
- Subirats J, Sharpe H, Santoro D, Topp E. 2023. Modeling antibiotic concentrations in the vicinity of antibiotic-producing bacteria at the micron scale. Applied and Environmental Microbiology. 89(4):e0026123. https://doi.org/10.1128/aem.00261-23 (en anglais seulement)
- Subirats J, Sharpe H, Tai V, Fruci M, Topp E. 2023. Metagenome meta-analysis reveals an increase in the abundance of some multidrug efflux pumps and mobile genetic elements in chemically polluted environments. Applied and Environmental Microbiology. 20:e01047-23. https://doi.org/10.1128/aem.01047-23 (en anglais seulement)
- Subirats J, Sharpe H, Topp E. 2022. Fate of Clostridia and other spore-forming Firmicute bacteria during feedstock anaerobic digestion and aerobic composting. Journal of Environmental Management 309: 114643. https://doi.org/10.1016/j.jenvman.2022.114643 (en anglais seulement)
- Tapp K, Deschênes M, Cooper A, Carrillo C, Blais B. 2023. Genomically informed custom selective enrichment of shiga toxigenic Escherichia coli (STEC) outbreak strains in foods using antibiotics. Journal of Food Protection 86(3):100052. https://doi.org/10.1016/j.jfp.2023.100052 (en anglais seulement)
- Tisza MJ, Smith DDN, Clark AE, Youn JH, NISC Comparative Sequencing Program, Khil PP, Dekker JP. 2023. Roving methyltransferases generate a mosaic epigenetic landscape and influence evolution in Bacteroides fragilis group. Nat Commun 14. https://doi.org/10.1038/s41467-023-39892-6 (en anglais seulement)
- VanderBurgt JT, Harper O, Garnham CP, Kohalmi SE, Menassa R. 2023. Plant production of a virus-like particle-based vaccine candidate against porcine reproductive and respiratory syndrome. Front. Plant Sci. 14:1044675. https://doi.org/10.3389/fpls.2023.1044675 (en anglais seulement)
- Venkatesan, M., Fruci, M, Verellen, LA, Skarina, T., Mesa, N., Flick, R., Stogios, P, and Savchenko A. 2023. Molecular mechanism of plasmid-borne resistance to sulfonamide antibiotics. Nature Communication. https://doi.org/10.1038/s41467-023-39778-7 (en anglais seulement)
- Waliaula, P. K., Kiarie, E. G. and Diarra M, S. 2024. Predisposition factors and control strategies of avian pathogenic Escherichia coli in laying hens. Front. Vet. Sci. 11:1474549. https://doi.10.3389/fvets.2024.1474549 (en anglais seulement).
- Yang C, Das Q, Rehman MA, Yin X, Shay J, Gauthier M, Lau CHF, Ross K, Diarra MS. 2023. Microbiome of Ceca from Broiler Chicken Vaccinated or Not against Coccidiosis and Fed Berry Pomaces. Microorganisms. 11:1184. https://doi.org/10.3390/microorganisms11051184 (en anglais seulement)
- Yao L, Cooper AL, Gill A, et al. 2024. Overcoming Microbial Inhibition of S. Sonnei Through the Exploitation of Genomically Predicted Antibiotic Resistance Profiles for the Development of Food Enrichment Media. Journal of Food Protection. 2024 Jul;87(7):100302. DOI: 10.1016/j.jfp.2024.100302. PMID: 38754553
- Yilmaz G, Chan M, Lau CH, Capitani S, Kang M, Charron P, Hoover E, Topp E, Guan J. 2024. How Gut Microbiome Perturbation Caused by Antibiotic Pre-Treatments Affected the Conjugative Transfer of Antimicrobial Resistance Genes. Microorganisms. 12(11):2148. doi: 10.3390/microorganisms12112148.
- Young KM, Isada MJ, Reist M, Uhland FC, Sherk LM, Carson CA. 2022. A scoping review of the distribution and frequency of extended-spectrum β-lactamase (ESBL)-producing Enterobacteriaceae in shrimp and salmon. Epidemiol Infect. 151:e1. https://doi.org/10.1017/S0950268822001819 (en anglais seulement)
- Zaidi SE, Zaheer R, Barbieri R, Cook SR, Hannon SJ, Booker CW, Church D, Van Domselaar G, Zovoilis A, McAllister TA. 2022. Genomic Characterization of Enterococcus hirae From Beef Cattle Feedlots and Associated Environmental Continuum. Front Microbiol. 13:859990. https://doi.org/10.3389/fmicb.2022.859990 (en anglais seulement)
- Zaidi SE, Zaheer R, Poulin-Laprade D, Scott A, Rehman MA, Diarra M, Topp E, Domselaar GV, Zovoilis A, McAllister TA. 2023. Comparative Genomic Analysis of Enterococci across Sectors of the One Health Continuum. Microorganisms. 11(3):727. https://doi.org/10.3390/microorganisms11030727 (en anglais seulement)
- Zaidi SE, Zaheer R, Thomas K, Abeysekara S, Haight T, Saville L, Stuart-Edwards M, Zovoilis A, McAllister TA. 2023. Genomic Characterization of Carbapenem-Resistant Bacteria from Beef Cattle Feedlots. Antibiotics (Basel, Switzerland), 12(6), 960. https://doi.org/10.3390/antibiotics12060960 (en anglais seulement)
- Zhang L, Said LB, Hervé N, Zirah S, Diarra MS, Fliss I. 2022. Effects of drinking water supplementation with Lactobacillus reuteri, and a mixture of reuterin and microcin J25 on the growth performance, caecal microbiota and selected metabolites of broiler chickens. Journal of Animal Science and Biotechnology. 13:34. https://doi.org/10.1186/s40104-022-00683-6 (en anglais seulement)
- Zhang L, Said LB, Diarra MS, Fliss I. 2022. Effects of bacterial derived antimicrobial solutions on shelf-life, microbiota and sensory attributes of raw chicken legs under refrigerated storage condition, International Journal of Food Microbiology 389: 109958. https://doi.org/10.1016/j.ijfoodmicro.2022.109958 (en anglais seulement)
Contactez-nous
L'Initiative de R-D en génomique
Courriel : info@grdi-irdg.collaboration.gc.ca